核酸的折叠热力学可以用自由能变、焓变和熵变等参数定量描述,这对于表征核酸结构的稳定性和功能性至关重要。预测和测量基于经典碱基配对规则的DNA结构的热力学特性相对容易,但对于在生物学功能和生物技术应用上同样十分重要的非经典核酸(G-四链体、三链体和i-基序等)而言,直接测量其在原位环境下的热力学仍然具有挑战性。
化学学院李峰教授课题组基于可编程的动态DNA化学,开发了自由能迁移分析(FESA)工具,成功实现了非经典核酸折叠热力学参数的原位表征。相较于传统的DNA热力学测量方法(DNA熔解或滴定),它具有以下主要优势:(1)FESA提供自由能转移的直接测量,不依赖于数学假设或近似,从而确保结果的可靠性。(2)它允许在原位条件下表征核酸结构,无需不断调整温度和反应物比等实验参数,可在各种温度、pH水平和金属离子浓度下有效运行。(3)FESA灵敏度高,可以在低纳摩尔浓度范围内测量微小的热力学变化。(4)FESA不需要专门的仪器,因此任何具有基本台式设施的生化实验室都可以使用。FESA的成功开发标志着通过分子工具表征非规范DNA结构热力学的技术进步,开辟了从分子工程和动态DNA化学视角探究基本化学和生物物理问题的新途径。
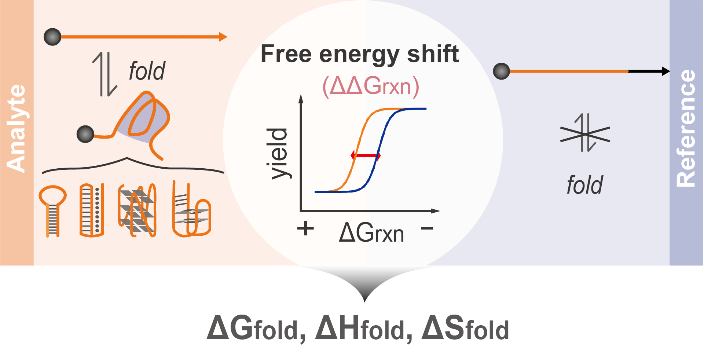
图1.FESA的原理示意图
该工作于2024年6月发表于Journal of the American Chemical Society期刊,题目为“Native characterization of noncanonical nucleic acid thermodynamics via programmable dynamic DNA chemistry”。四川大学化学学院为第一单位,李峰教授为通讯作者,化学学院博士研究生吴宇琴和专职博士后王冠为共同第一作者。
文章链接:https://doi.org/10.1021/jacs.4c04721

